1. Cargar los paquetes y la tabla de datos.
Primero cargamos el paquete “bipartite”, si aun no esta instalado debemos instalarlo primero con la funcion install.packages(“bipartite”)
library(bipartite)
data("bezerra2009")En este ejemplo usaremos la tabla 1 de datos de bezerra2009 la cual consta de observaciones de 38 individuos pertenecientes a 13 especies de plantas oleaginosas de la familia Malphighiaceae. Se colectaron los visitantes florales y solo se consideraron los visitantes legítimos. Los números en la tabla se refieren a la cantidad de visitantes de cada especie de abeja recolectada en cada especie de planta. El número de abeja que visitaron las flores se registraron durante cuatro días consecutivos, de 5.00 a 17.00 con un total de 1392h de observaciones.
Ubicación: Parque Nacional do Catimbau, Brasil (8°24’00" -37°36’35“S y 3°09’30” -37°14’40"W)
Para mas información puede visitar http://www.nceas.ucsb.edu/interactionweb
| Centris.aenea | Centris.fuscata | Centris.caxiensis | Centris.tarsata | Centris.flavifrons | Centris.trigonoides | Centris.obsoleta | Epicharis.sp2 | Apis.mellifera | Centris.sp3 | Centris.sp1 | Xylocopa.sp | Xylocopa.grisescens | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diplopterys.pubipetala | 1368 | 1364 | 740 | 460 | 416 | 256 | 0 | 328 | 364 | 368 | 0 | 0 | 0 |
| Byrsonima.gardnerana | 924 | 320 | 2108 | 464 | 284 | 0 | 0 | 0 | 28 | 0 | 0 | 0 | 0 |
| Banisteriopsis.muricata | 396 | 468 | 108 | 140 | 272 | 652 | 912 | 364 | 44 | 0 | 368 | 164 | 84 |
| Heteropterys.sp1 | 764 | 680 | 528 | 308 | 404 | 300 | 0 | 28 | 0 | 0 | 0 | 76 | 0 |
| Heteropterys.sp2 | 740 | 656 | 528 | 332 | 0 | 324 | 0 | 116 | 116 | 0 | 0 | 0 | 0 |
| Dicella.bracteosa | 556 | 512 | 356 | 132 | 524 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Carolus.chasei | 604 | 452 | 432 | 200 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Stigmaphyllon.paralias | 504 | 816 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Banisteriopsis.stellaris | 292 | 300 | 244 | 116 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Banisteriopsis.schizoptera | 228 | 224 | 124 | 120 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Stigmaphyllon.auriculatum | 240 | 164 | 68 | 0 | 196 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Stigmaphyllon.ciliatum | 268 | 196 | 0 | 0 | 164 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
| Janusia.anisandra | 188 | 244 | 0 | 96 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 | 0 |
2. Graficar
Cuando los datos no se encuentren organizados de mayor a menor se debe usar antes la funcion sortweb() sobre los datos
plotweb(bezerra2009, method="normal", text.rot="90",
labsize=0.9, col.low="#CDCD00", col.high="#27408B", col.interaction="cornsilk4")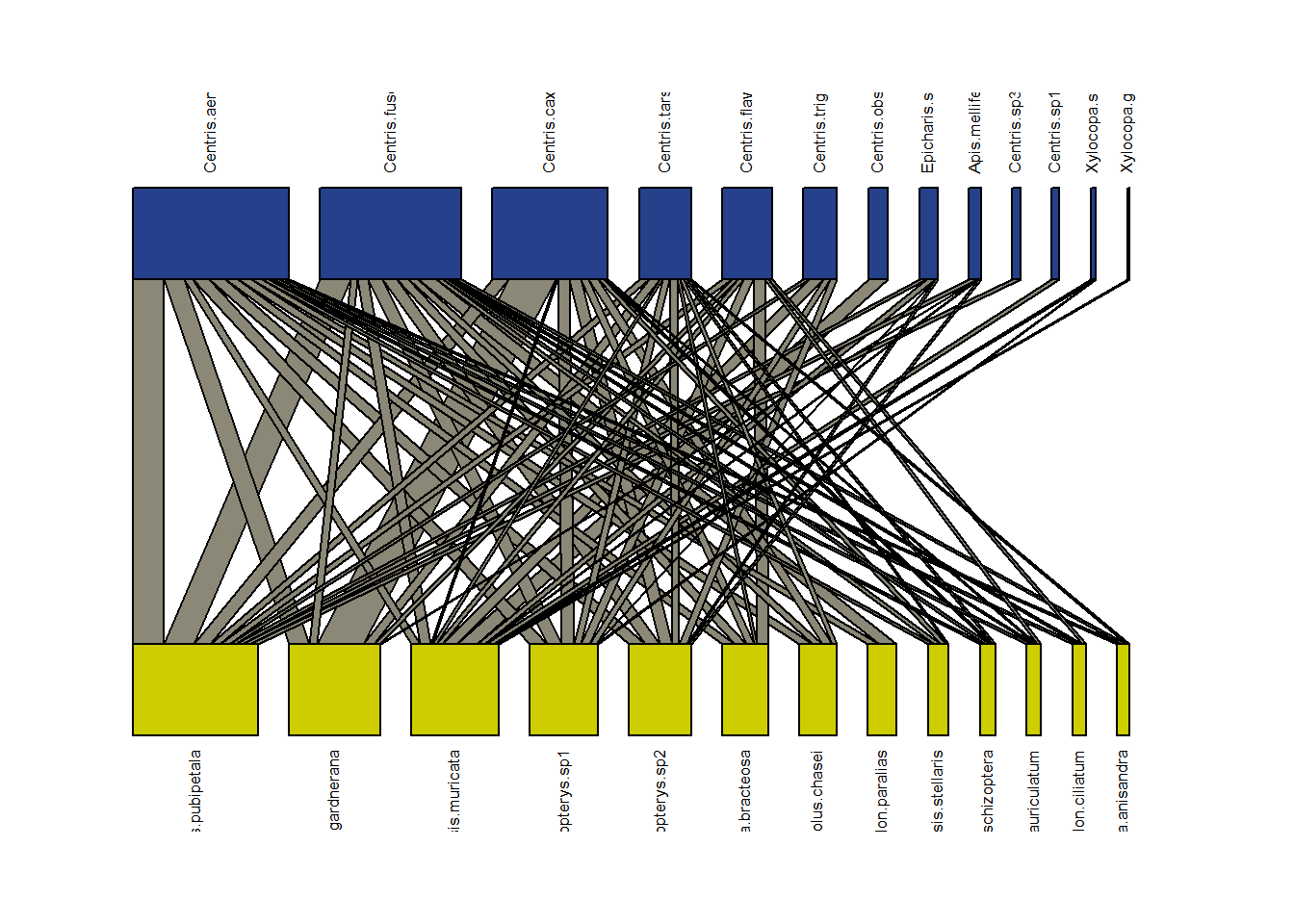
Estas interacciones tambien se pueden graficar de otra manera a traves de la funcion visweb()
visweb(bezerra2009, plotsize=9)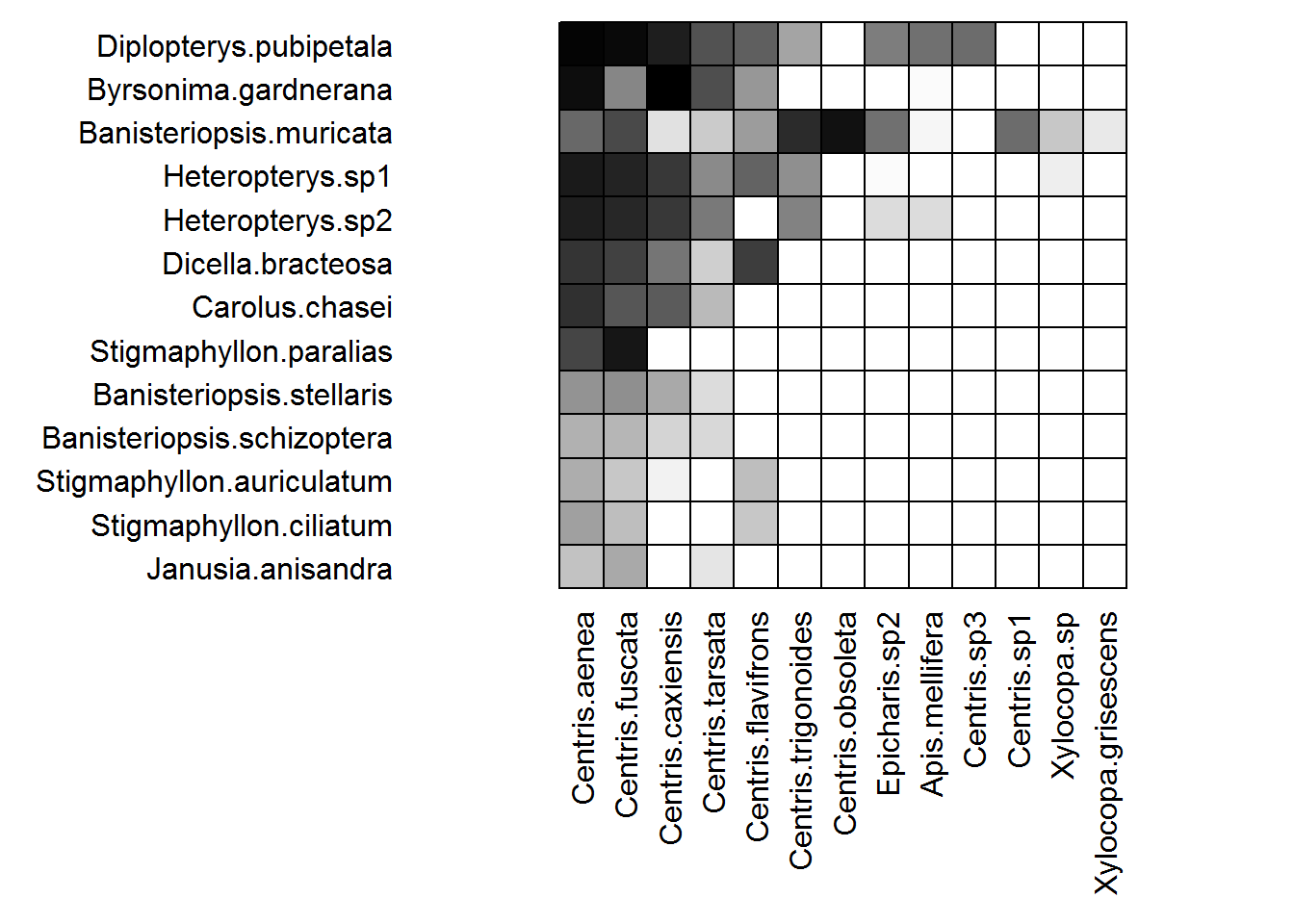
Usando el paquete NetworkD3
Paquetes necesarios
library(networkD3)
library(tidyverse)
library(bipartite)1. Convertimos la matrix de datos a un Data Frame
datos <- bezerra2009 %>% as_tibble(rownames = "source")2. Covertimos la tabla a la forma adecuada para ser leida por el paquete
links <- datos %>%
pivot_longer(2:14, names_to = "target", values_to = "value")3. Creamos un Data Frame con todas especies de plantas
nodes <- data.frame(
name=c(as.character(links$source),
as.character(links$target)) %>% unique()
)4. Hacer las conecciones con un ID.
links$IDsource <- match(links$source, nodes$name)-1
links$IDtarget <- match(links$target, nodes$name)-15. Hacer el plot
sankeyNetwork(Links = links, Nodes = nodes,
Source = "IDsource", Target = "IDtarget",
Value = "value", NodeID = "name",
sinksRight=F, fontSize = 10)Usando el paquete circlize
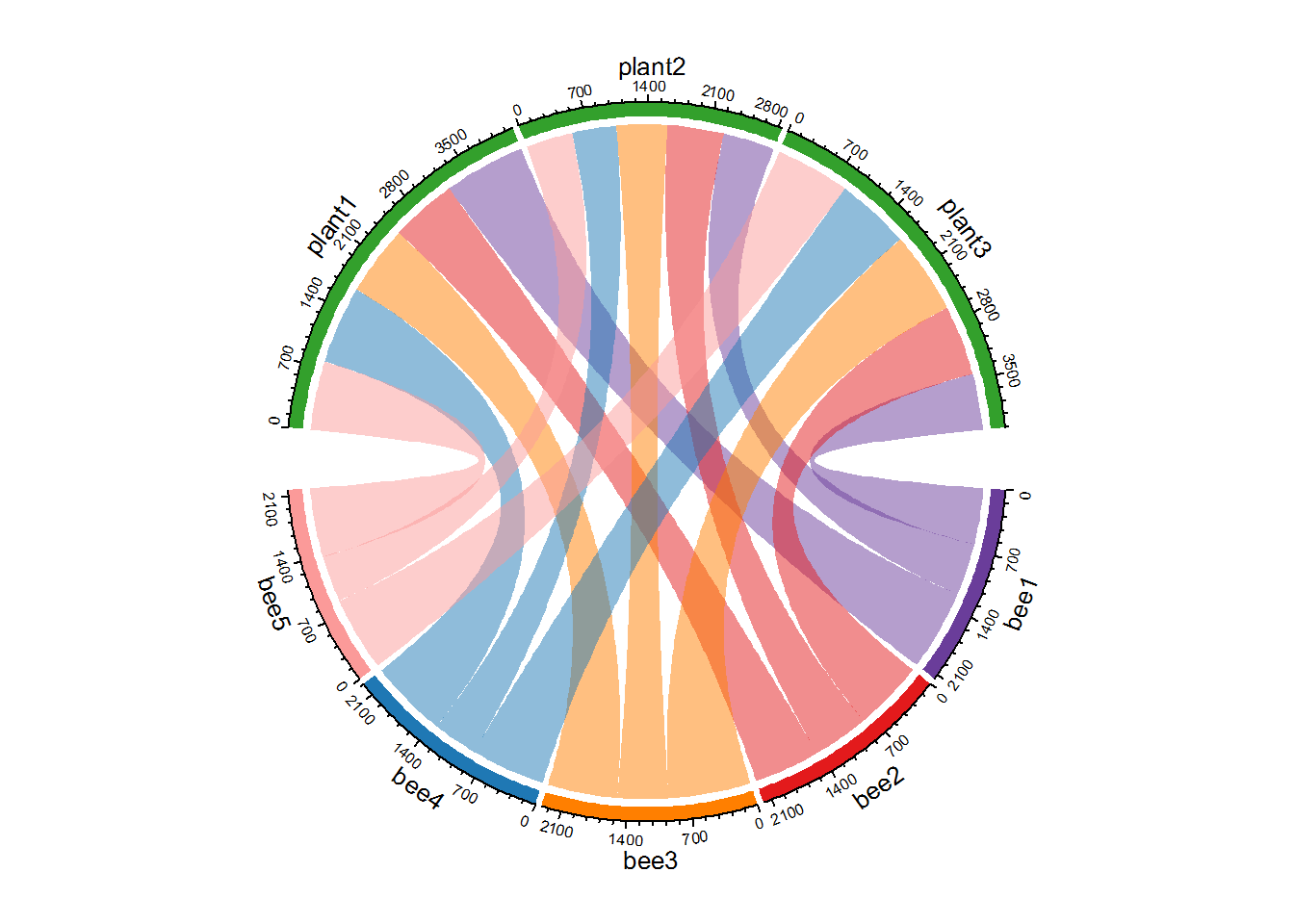
Paquetes necesarios
library(circlize)
library(tidyverse)1. Creamos una matrix de datos
valores <- c(500:1000)
muestra <- sample(valores, 15)
col_pal <- c(bee1 = "#6a3d9a", bee2 = "#e31a1c", bee3 = "#ff7f00", bee4 = "#1f78b4", bee5 = "#fb9a99",
plant1 = "#33a02c", plant2 = "#33a02c" , plant3 = "#33a02c")
tabla <- matrix(muestra, nrow = 5, dimnames = list(c("bee1", "bee2", "bee3", "bee4", "bee5"),
c("plant1", "plant2", "plant3")))2. Graficamos
chordDiagram(tabla, grid.col = col_pal)